ヒストグラムと密度
データをヒストグラム化するときにつまづいたのでメモ。
import numpy as np import matplotlib.pyplot as plt np.random.seed(0) data = np.random.randn(1000) plt.hist(data, bins=10, color='red' , alpha=0.5) plt.hist(data, bins=30, color='blue', alpha=0.5) plt.show()
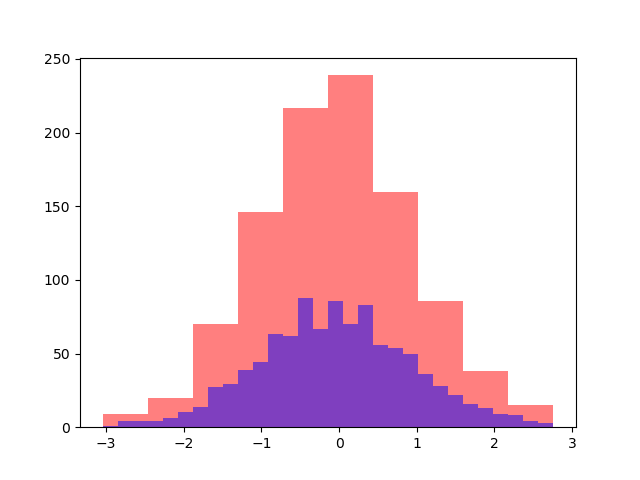
matplotlib のヒストグラムはデフォルトでは個数を表示する。このため bins の数を増やすと、ひとつの bin あたりの個数が減るため、全体の面積が減少する。これは当たり前のことで、解決するためには density=True にする。
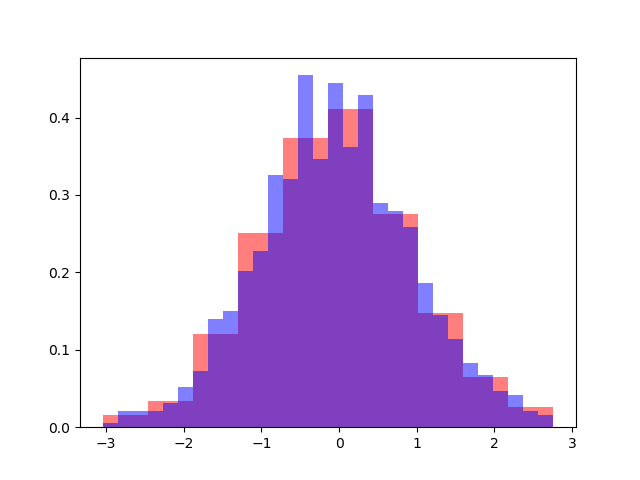
まあこれは当たり前なんだが、先日わけあってヒストグラムの幅をランダムにするということをしたところ、density を使わないと同じデータにも関わらず全く概形の異なるヒストグラムができてしまうことに気づいた。
import numpy as np import matplotlib.pyplot as plt np.random.seed(1) data = np.random.randn(1000) bins = np.random.random(31) * 6 - 3 bins.sort() print(bins) plt.hist(data, bins=bins, color='red' , alpha=0.5, density=True) plt.hist(data, bins=30, color='blue', alpha=0.5, density=True) plt.show()
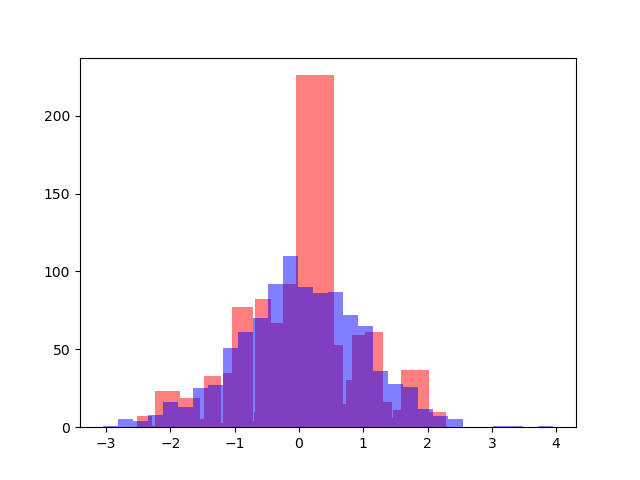
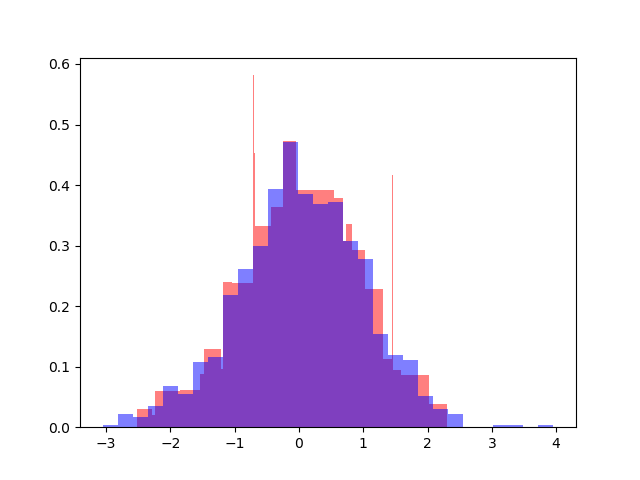
あるデータが理論的な確率分布とどれだけ一致するかを調べる時は density を使わないといけない、というきわめて初歩的なミスを犯した。
Intel TBB による並列化
とりあえずこういうシンプルな for 構文を並列化したい。
#include <iostream> #include <vector> int main () { const int N = 100; std::vector<int> v(N); for(int i=0; i<N; ++i) { v[i] = i*i; } }
まず for の中身を lambda 式に書き換える。
#include <iostream> #include <vector> int main () { const int N = 100; std::vector<int> v(N); auto calc = [&](int i) { v[i] = i*i; }; for(int i=0; i<N; ++i) { calc(i); } }
続いてこれを tbb にする。
#include <iostream> #include <vector> #include <tbb/parallel_for.h> #include <tbb/task_scheduler_init.h> int main () { const int N = 100; std::vector<int> v(N); auto calc = [&](int i) { v[i] = i*i; }; tbb::parallel_for(size_t{0}, (size_t)N, calc); }
コンパイルするときは -ltbb オプションをつける。
C++/Eigen での RMSD計算
タンパク質の構造変化を定量化する概念として RMSD (Root mean square deviation) というのがある。これは要するに距離の差の二乗平均なので非常に汎用性がありそうなのだが、タンパク質の構造以外の用途に使う人を見たことがない。
Biopython に SVD (特異値分解) を使って RMSD を計算するコードがあったので、これを参考に C++ Eigen における RMSD 計算のコードを書いた。
タンパク質の原子座標には構造変化のほかに並進や回転の自由度もあるので、これをあらかじめ除去し、構造変化による部分だけのRMSDを計算する。手順は以下のとおり。
- 座標を N×3 行列に格納する (N は原子数)
- 両構造の重心を原点に合わせる
- 特異値分解で回転行列を求める
- 回転を実行しRMSDを計算する
という手順。
#include <iostream> #include <Eigen/SVD> #include <Eigen/LU> using Mat = Eigen::Matrix<double, Eigen::Dynamic, 3>; using SqMat = Eigen::Matrix3d; int main() { Mat ref, tar; // reference, target // 座標入力は省略 // 重心を原点へ Eigen::Vector3d centroid; centroid = ref.colwise().sum() / ref.rows(); ref.rowwise() -= centroid.transpose(); centroid = tar.colwise().sum() / tar.rows(); tar.rowwise() -= centroid.transpose(); // SVD をとる SqMat a, U, V, rot; a = tar.transpose() * ref; Eigen::JacobiSVD<SqMat> svd(a, Eigen::ComputeFullU | Eigen::ComputeFullV); U = svd.matrixU(); V = svd.matrixV(); rot = (V*U.transpose()).transpose(); // 鏡像チェック if (rot.determinant() < 0) { V.row(2) *= -1; rot = (V*U.transpose()).transpose(); } // ref のうえに superimpose する Mat tar_on_ref = tar * rot; // RMSD を計算 Mat diff = tar_on_ref - ref; double rmsd = std::sqrt((diff.array() * diff.array() / diff.rows()).sum()); std::cout << "rmsd: " << rmsd << '\n'; }
Eigen で重心
C++ の線形代数ライブラリ Eigen で、3次元位置ベクトルを10個並べた Matrix があって、これの重心が原点になるようにしたい場合
Eigen::Matrix<double, 10, 3> position; // 座標代入部分省略 // 重心ベクトル Eigen::Vector3d centroid; centroid = position.colwise().sum() / position.rows(); ref.rowwise() -= centroid.transpose();
という具合になる。python/numpy 系に比べると colwise, rowwise といった少々直感的にわかりづらいものを使う必要がある。
enum を for で回す
enum の最後に DUMMY を追加して、int 型にキャストすれば for で回せる。もっといいやり方がありそうな気がする。
#include <iostream> enum Week { Sun ,Mon ,Tue ,Wed ,Thu ,Fri ,Sat ,DUMMY }; int main() { for(int i=0; i<static_cast<int>(DUMMY); i++) { std::cout << static_cast<Week>(i) << '\n'; } }
実行結果
0 1 2 3 4 5 6
std::vector を mutex でロックしてマルチスレッドで書き込む
C++ の std::thread でひとつの vector に push_back しまくるコード
#include <iostream> #include <vector> #include <thread> int main() { std::vector<int> vi; std::vector<std::thread> vt; for(int i=0; i<100; ++i) { vt.push_back(std::thread([&i, &vi]{ vi.push_back(i); })); } for(int i=0; i<100; ++i) { vt[i].join(); } return 0; }
これをコンパイルして実行すると
$ g++ test.cpp -pthread $ ./a.out (...) Aborted (core dumped)
当然こうなる。 というわけで mutex を使って vector をロックするようにする。正確には、vector をメンバとして持ち、ロックもできる class をつくる。
#include <iostream> #include <vector> #include <thread> #include <mutex> class myvector { std::mutex mtx; std::vector<int> data; public: void push_back(int x) { std::lock_guard<std::mutex> lock(mtx); data.push_back(x); } void print() { for(int x: data) { std::cout << x << '\n'; } } }; int main() { const int N = 100; std::vector<std::thread> vt; myvector mv; for(int i=0; i<N; i++) { vt.push_back(std::thread([=, &mv]{ mv.push_back(i); })); } for(int i=0; i<N; i++) { vt[i].join(); } mv.print(); }
これで 0 〜 99 までの数字が出力される。順番が正確に 0 〜 99 ではないのは並列化のため。
なお最初ラムダ式のところをうっかり
vt.push_back(std::thread([&i, &mv]{ mv.push_back(i); }));
と書いていたが、これだと i がコピーではなく参照になってしまうので、 mv に入る i の値が正確に 0〜99 でなくなってしまった(たいてい 2 から始まる)。ラムダ式に対し意味もわからず「使う変数を & で書いとくんやろ」という甘い考えを持っていたことを反省。